近日,武汉大学病毒学国家重点实验室严欢研究组在国际知名期刊 Nature Communications 在线发表题为“Sarbecovirus RBD indels and specific residues dictating multi-species ACE2 adaptiveness”的研究论文。该研究系统分析了沙贝病毒(Sarbecoviruses亚属病毒,即SARS相关冠状病毒)刺突蛋白受体结合基序(RBM)中插入/缺失(indels)等序列特征与其识别多物种ACE2受体能力之间的关系,揭示了沙贝病毒在适应ACE2中三条不同的进化路径,为科学评估沙贝病毒跨种传播能力和潜在人兽共患风险提供了重要参考。

2002年与2019年暴发的非典与新冠疫情给全球公共卫生与经济发展带来了巨大冲击,引发了人们对沙贝病毒潜在人兽共患病风险的高度关注。尽管已有许多研究揭示这些病毒识别ACE2受体的机制,对于不同沙贝病毒识别多物种ACE2受体的能力差异,及其分子机制仍不清晰,尤其是近年来报道的多种具有RBM插入/缺失的沙贝病毒。由于众多沙贝病毒仅仅以序列的形式为人们所知,并未分离获得毒株,能否通过分析病毒刺突蛋白序列特征预测这些病毒对人类和其他哺乳动物的威胁具有重要意义。
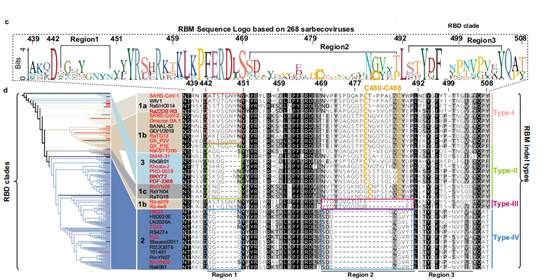
图1 根据沙贝病毒RBM中的插入缺失特征将其分为四种RBM indel类型
在该研究中,严欢团队通过对已公开的268条沙贝病毒刺突蛋白序列进行细致分析,提出了一种新的分类方法,即根据其RBM序列缺失特征将其分为四种RBM indel类型(type I-typeIV)(图1)。研究人员通过RBD结合和假病毒入侵等功能实验,评估了20种代表性病毒及其突变株利用51种蝙蝠和5种其他哺乳动物ACE2受体的能力。
结果表明:拥有较长RBM(Type I)的病毒通常有能力识别多物种ACE2,RBM中含有一段缺失(Type II和Type III)的病毒仅能识别较少数物种(如宿主)的ACE2,而RBM两个区域均存在片段缺失的沙贝病毒(Type IV)则未被发现有任何利用ACE2的能力。更为深入的分析揭示,存在RBM片段缺失的病毒适应多物种ACE2的能力相对受限,而在相互作用关键位点的氨基酸差异进一步调节了其ACE2识别谱的宽窄(图2)。值得一提的是,仅在Type IV沙贝病毒(两段缺失)中补齐RBM的缺失序列仍不能使病毒获得任何使用ACE2的能力,原因在于RBM之外的某些具有clade2谱系特异性的残基限制了它们利用ACE2受体的能力,进一步支持这些病毒已适应了另一种未知受体的假说。

图2. 沙贝病毒四种RBM indel 类型及其ACE2 适应能力
该研究不仅为深入理解沙贝病毒的ACE2识别机制提供了重要证据,还提出了RBM插入/缺失在病毒进化中调节多物种ACE2适应性的新见解。根据该假说,非洲与欧洲沙贝病毒中存在的RBM Region1的缺失可能是祖先病毒的特征,但病毒在亚洲地区进化过程中,分别朝着维持现状、高度适应和受体转换三条不同路径进化。这些序列插入缺失、关键残基变异、二硫键以及糖基化修饰的不同特征共同塑造了沙贝病毒利用多物种ACE2的能力,进而影响其跨种传播的潜力(图3)。综上,该研究系统性绘制了沙贝病毒多物种ACE2受体的识别谱,深入探讨了影响沙贝病毒跨种传播潜力的关键因素,为预测未来潜在的冠状病毒大流行提供了重要的科学依据。
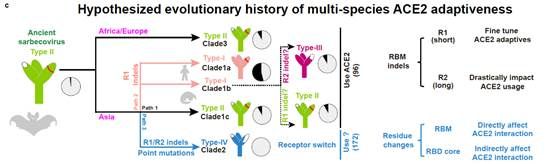
图3. 沙贝病毒ACE2识别能力的三种不同进化方向
武汉大学病毒学国家重点实验室严欢教授为该论文的通讯作者,武汉大学生命科学学院博士研究生司竣宇、硕士研究生陈媛梅、孙晔晖为共同第一作者。中科院武汉病毒研究所石正丽研究员为本工作提供了宝贵意见。该研究得到了武汉大学泰康生命医学中心以及国家重点研发计划、国家自然科学基金等项目的资助。
原文链接:https://www.nature.com/articles/s41467-024-53029-3