由新冠病毒(SARS-CoV-2)引起的疫情仍在世界各国肆虐。据最新统计数据显示,截止2021年3月,全球新冠肺炎累计确诊病例数超1.1528亿例,累计死亡超255万例。新冠疫情的防控、理解新冠RNA感染和复制等分子机制、抗病毒药物及疫苗的开发仍是当下的主要任务。
2021年3月4日,武汉大学病毒学国家重点实验室周宇、陈宇及蓝柯团队合作在知名学术期刊-Molecular Cell在线发表题为 “The SARS-CoV-2 Subgenome Landscape and its Novel Regulatory Features”的研究论文。该研究结合Nanopore、 NGS测序技术及传统研究手段,利用多种新冠病毒细胞感染模型构建了新冠病毒亚基因组的时序动态图谱,发现了在病毒亚基因组生成过程中的新模式,并揭示了介导新冠病毒亚基因组生成中的RNA-RNA互作规律及关键特征(图1)。为理解新冠病毒的感染与复制过程以及抗病毒药物和疫苗开发提供了新的线索。

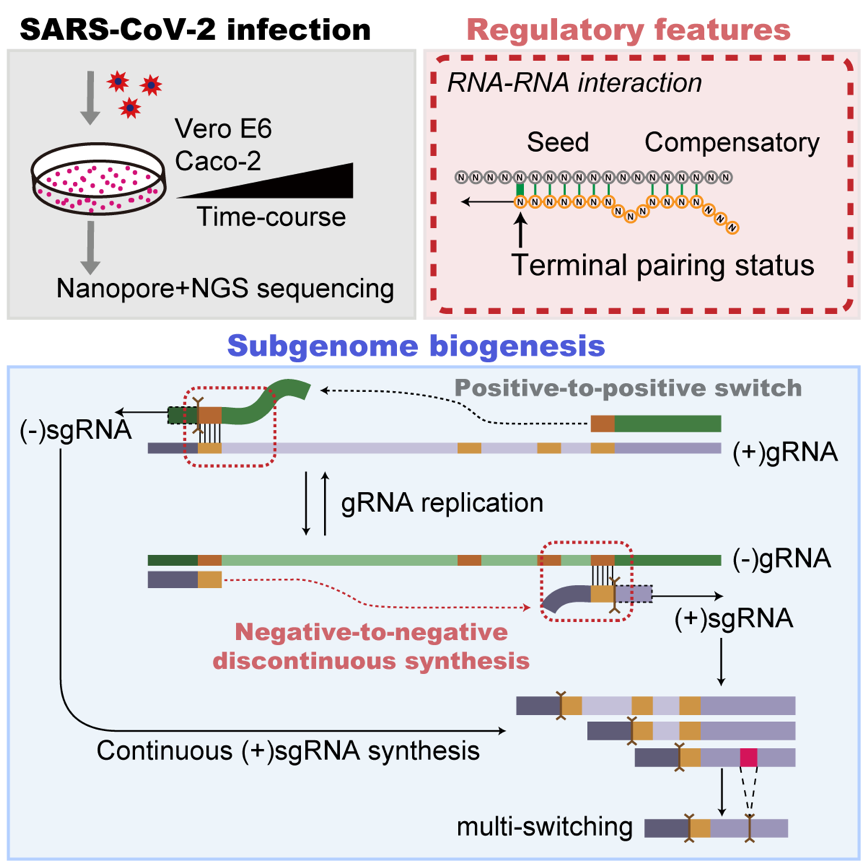
图1 实验策略、新冠病毒亚基因组生成模式及调控特征概览
新冠病毒是一种单股正链RNA包膜病毒,基因组长约30,000 nt。之前的研究显示,冠状病毒以正链RNA基因组为模板利用RdRP合成负链RNA。其中,连续合成可产生互补的全长负链基因组RNA,而不连续合成则通过模板跳跃产生不同长度的负链亚基因组RNA (sgRNA)。这些负链RNA中间体作为模板可经由RdRP合成正链子代基因组RNA和亚基因组RNA,由此完成基因组的复制和转录过程。生成亚基因组RNA过程中的模板跳跃及不连续合成称为“模板转换”(template switch),这是病毒高效生成可翻译RNA的途径。
在本研究中,该团队利用2005年研究SARS冠状病毒亚基因组复制转录的经验及传统手段,进一步整合Nanopore长读长测序与NGS深度测序技术及生物信息学分析,建立了绿猴Vero E6细胞系和人类Caco-2细胞系在感染新冠病毒后不同时间点的病毒亚基因组动态图谱,鉴定出7499个template switch位点,并重构出433条(208个cluster)高置信度的全长亚基因组序列。定量分析发现新冠病毒亚基因组中multi-switching(一个亚基因组中出现两个或两个以上的template switch事件)现象中的多个模板转换事件可级联发生。
该研究进一步探索了控制亚基因组生成效率的调控特征。通过分析介导亚基因组生成的RNA互作模式和配对能量,发现新冠病毒在亚基因组生成过程中除了经典的“positive-to-positive template switch”模式(正链模板合成负链sgRNA过程中发生的template switch),也有部分亚基因组是由“negative-to-negative template switch” 模式(负链模板合成正链sgRNA过程中发生template switch)产生的。并发现新冠病毒不同亚基因组sgRNAs的丰度与template switch位点周围RNA-RNA配对能量、连续配对长度,特别是末端配对状态相关(图2)。以前的template switch模型认为,特定的TRS-motif介导了template switch的发生。该研究发现有相当种类的亚基因组缺少TRS-motif支持,揭示更加泛化的RNA-RNA互作而非特异TRS-motif是调控病毒亚基因组RNA生成的分子基础。
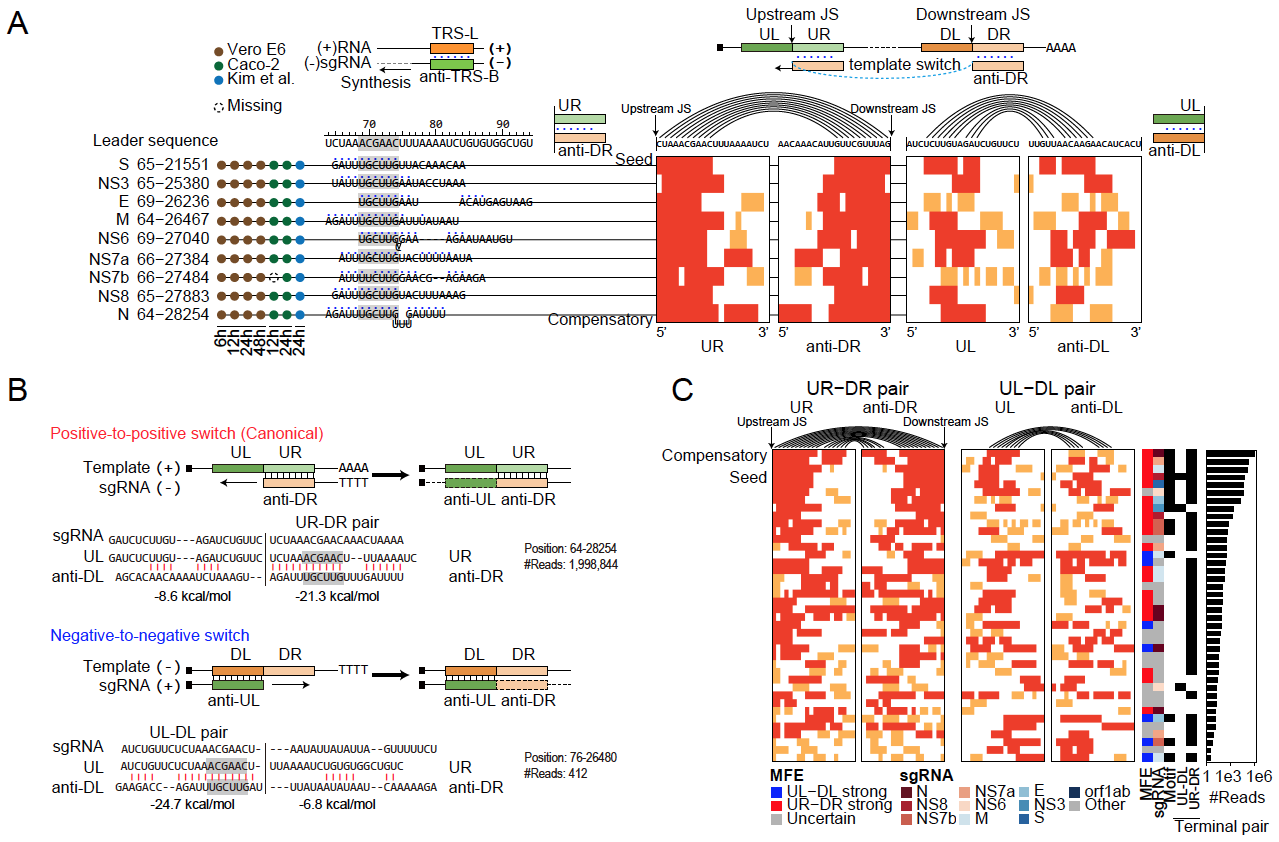
图2 解析新冠病毒亚基因组生成调控规律示意图
此外,该研究发现新冠病毒在ORF1ab基因与N基因区域存在分布广泛、种类多但丰度较低的非经典template switch事件(ORF1ab类型),该现象在新冠病毒感染的不同样品中均存在。通过对公开的新冠病毒感染的Ribo-seq和质谱数据分析,发现部分ORF1ab类型亚基因组可能拥有编码新蛋白/肽段的潜力。这一研究比较全面地揭示了新冠病毒亚基因组sgRNA的生成模式和调控规律,为开发新的抗病毒药物提供了潜在的靶点和新思路。
周宇教授、陈宇教授与蓝柯教授为该论文的共同通讯作者,武汉大学生命科学学院博士生王得和、江翱、冯姜鹏、李广楠和郭冬为该论文共同第一作者,中科院武汉病毒所石正丽研究员、杨兴娄研究员、武汉大学陈明周教授、郭铭老师、博士生刘乾运等参与了本研究。研究工作得到了武汉大学动物三级生物安全实验室的大力支持以及武汉大学新冠科技攻关专项基金、北京泰康溢彩公益基金会、国家自然科学基金新冠病毒专项等资助。
原文链接:
https://www.cell.com/molecular-cell/fulltext/S1097-2765(21)00166-0